決定木¶
このセクションで論じられる ML クラスは,分類木および回帰木のアルゴリズムの実装であり,これについては [Breiman84] で説明されています.
CvDTree クラスは,単独で利用できる単一の決定木,または決定木アンサンブル( Boosting と Random Trees を参照してください)の基底クラスを表現します.
決定木は二分木(つまり,葉以外の各ノードがちょうど2つずつの子ノードを持つ木)です.それぞれの葉がクラスラベルを持つ(複数の葉が同じラベルを持つこともあります)場合には,分類に利用できます.また,それぞれの葉が定数を持つ場合には,回帰に利用できます(このような決定木は近似関数とも見なせますが,これは区分的に一定な値(つまり離散値)を出力することになります).
決定木による予測¶
葉ノードに到達し,そこで入力特徴ベクトルに対する応答を得るために,根(ルート)ノードから予測手続きが開始されます. この手続きでは,特定の変数に基づいて,葉以外の各ノードから左(つまり次の観測ノードとして左側の子ノードを選択),あるいは右に分岐して進み,そのインデックスは観測ノードに保存されます. この変数は,連続変数またはカテゴリ変数です. 1番目(連続変数)の場合,入力された変数値が特定の閾値(これもノードに保存されています)と比較され,その値が閾値より小さい場合は左に,そうでない場合は右に進みます(例えば,重量が 1kgよりも軽い場合は左に,重い場合は右に進みます). そして,2番目(カテゴリ変数)の場合,入力された離散変数値が,それが取りうる値の有限集合のうち,特定の部分集合(これもノードに保存されています)に属するか否かが調べられます. そして,属している場合は左に,そうでない場合は右に進みます(例えば,色が緑か赤の場合は左に進み,そうでない場合は右に進みます). つまり,各ノードで1つのエンティティペア(variable _ index, decision _ rule(閾値/部分集合))が利用されます. このペアは,分岐(変数 variable _ index による分岐)と呼ばれます.葉ノードに到達した時点で,そのノードに割り当てられていた値が予測手続きの出力値となります.
たまに,入力ベクトルのある特徴が欠損する(例えば,暗い場所ではオブジェクトの色を測定することが困難)と,予測手続きが,あるノード(前述の例では色で,分岐するノードの条件)で行き詰まるかもしれません. このような状況を回避するために,決定木は,いわゆる代理分岐(surrogate split)を利用します.つまり最適な「第一」分岐に加えて,決定木の各ノードが,ほぼ同じ結果を与える1つ以上の別の変数による分岐を持つことがあります.
決定木の学習¶
決定木は,根ノードから再帰的に構築されます.根ノードの分岐には,すべての学習データ(特徴ベクトル,およびそれに対する応答)が用いられます.各ノードにおいて,ある基準(MLでは,分類には ジニ 「純粋」指標,回帰には誤差の2乗の和が用いられます)に基づいて,最適決定規則(つまり,最適な「第一」分岐)が求められます.また必要ならば,学習データにおける第一分岐の結果に類似した代理分岐が求められます.すべてのデータは,第一分岐と代理分岐によって(まさに予測手続きでされるように)左右の子ノードに分割されます.そして,この分割処理は左右のノードでも再帰的に行われます.以下のいずれかの場合に,各ノードにおける再帰的処理が停止します(つまり,ノードがそれ以上分岐しません):
- 構築された決定木の枝の深さが,指定した最大値に到達した場合.
- ノードの学習サンプル数が指定した閾値以下,つまり,そのサンプルがノードをさらに分岐させるための統計的な代表集合となり得ない場合.
- ノードの全サンプルが同じクラスに属する(あるいは回帰の場合,分散が非常に小さい)場合.
- その第一分岐が,単なるランダム選択と同程度の効果しか示さない場合.
木を構成する際に,必要ならば交差検証法により枝刈りが行われます.つまり,モデルを過剰適合させてしまうような木の枝が刈られます.この手続きは通常,単一で動作する決定木に対してのみ用いられます.一方,決定木アンサンブルの場合は通常,十分に小さい木々を構成することで,過剰適合に対する自身の防護機構が働きます.
変数の重要度¶
決定木は,予測はもちろんのこと,他にも様々なデータ解析に利用できます.ここで鍵となる決定木の性質の1つは,各変数の重要度(相対的な決定力)が計算できるということです.例えばスパムフィルタで,文章中に登場する単語の集合を特徴ベクトルとして用いると,変数の重要度評価は,最も「スパムらしい」単語を決定するために利用でき,その結果,適切な辞書のサイズを保つことができます.
各変数の重要度は,その変数が通過する木の第一分岐と代理分岐をすべて利用して計算されます.したがって,変数の重要度を正しく計算するためには,たとえデータ欠損がない場合でも,学習パラメータにおいて代理分岐が有効でなければいけません.
[Breiman84] Breiman, L., Friedman, J. Olshen, R. and Stone, C. (1984), “Classification and Regression Trees”, Wadsworth.
CvDTreeSplit¶
- CvDTreeSplit¶
決定木ノード分岐.
struct CvDTreeSplit
{
int var_idx;
int inversed;
float quality;
CvDTreeSplit* next;
union
{
int subset[2];
struct
{
float c;
int split_point;
}
ord;
};
};
CvDTreeNode¶
- CvDTreeNode¶
決定木ノード.
struct CvDTreeNode
{
int class_idx;
int Tn;
double value;
CvDTreeNode* parent;
CvDTreeNode* left;
CvDTreeNode* right;
CvDTreeSplit* split;
int sample_count;
int depth;
...
};
CvDTreeNode のその他多数のフィールドは,学習時に内部的に利用されます.
CvDTreeParams¶
- CvDTreeParams¶
決定木の学習パラメータ.
struct CvDTreeParams
{
int max_categories;
int max_depth;
int min_sample_count;
int cv_folds;
bool use_surrogates;
bool use_1se_rule;
bool truncate_pruned_tree;
float regression_accuracy;
const float* priors;
CvDTreeParams() : max_categories(10), max_depth(INT_MAX), min_sample_count(10),
cv_folds(10), use_surrogates(true), use_1se_rule(true),
truncate_pruned_tree(true), regression_accuracy(0.01f), priors(0)
{}
CvDTreeParams( int _max_depth, int _min_sample_count,
float _regression_accuracy, bool _use_surrogates,
int _max_categories, int _cv_folds,
bool _use_1se_rule, bool _truncate_pruned_tree,
const float* _priors );
};
この構造体は,決定木の学習パラメータをすべて含んでいます.デフォルトコンストラクタは,単独動作可能な分類木用に調整されたデフォルト値を用いて,すべてのパラメータを初期化します.どのパラメータもオーバーライド可能です,また,この構造体自体も,より高度なコンストラクタにより完全に初期化される可能性があります.
CvDTreeTrainData¶
- CvDTreeTrainData¶
決定木の学習データ,および決定木アンサンブル用の共有データ.
struct CvDTreeTrainData
{
CvDTreeTrainData();
CvDTreeTrainData( const CvMat* _train_data, int _tflag,
const CvMat* _responses, const CvMat* _var_idx=0,
const CvMat* _sample_idx=0, const CvMat* _var_type=0,
const CvMat* _missing_mask=0,
const CvDTreeParams& _params=CvDTreeParams(),
bool _shared=false, bool _add_labels=false );
virtual ~CvDTreeTrainData();
virtual void set_data( const CvMat* _train_data, int _tflag,
const CvMat* _responses, const CvMat* _var_idx=0,
const CvMat* _sample_idx=0, const CvMat* _var_type=0,
const CvMat* _missing_mask=0,
const CvDTreeParams& _params=CvDTreeParams(),
bool _shared=false, bool _add_labels=false,
bool _update_data=false );
virtual void get_vectors( const CvMat* _subsample_idx,
float* values, uchar* missing, float* responses,
bool get_class_idx=false );
virtual CvDTreeNode* subsample_data( const CvMat* _subsample_idx );
virtual void write_params( CvFileStorage* fs );
virtual void read_params( CvFileStorage* fs, CvFileNode* node );
// すべてのデータを解放します.
virtual void clear();
int get_num_classes() const;
int get_var_type(int vi) const;
int get_work_var_count() const;
virtual int* get_class_labels( CvDTreeNode* n );
virtual float* get_ord_responses( CvDTreeNode* n );
virtual int* get_labels( CvDTreeNode* n );
virtual int* get_cat_var_data( CvDTreeNode* n, int vi );
virtual CvPair32s32f* get_ord_var_data( CvDTreeNode* n, int vi );
virtual int get_child_buf_idx( CvDTreeNode* n );
////////////////////////////////////
virtual bool set_params( const CvDTreeParams& params );
virtual CvDTreeNode* new_node( CvDTreeNode* parent, int count,
int storage_idx, int offset );
virtual CvDTreeSplit* new_split_ord( int vi, float cmp_val,
int split_point, int inversed, float quality );
virtual CvDTreeSplit* new_split_cat( int vi, float quality );
virtual void free_node_data( CvDTreeNode* node );
virtual void free_train_data();
virtual void free_node( CvDTreeNode* node );
int sample_count, var_all, var_count, max_c_count;
int ord_var_count, cat_var_count;
bool have_labels, have_priors;
bool is_classifier;
int buf_count, buf_size;
bool shared;
CvMat* cat_count;
CvMat* cat_ofs;
CvMat* cat_map;
CvMat* counts;
CvMat* buf;
CvMat* direction;
CvMat* split_buf;
CvMat* var_idx;
CvMat* var_type; // i 番目の要素 =
// k<0 - 連続変数
// k>=0 - カテゴリ変数,配列 cat_* の k 番目の要素を参照します
CvMat* priors;
CvDTreeParams params;
CvMemStorage* tree_storage;
CvMemStorage* temp_storage;
CvDTreeNode* data_root;
CvSet* node_heap;
CvSet* split_heap;
CvSet* cv_heap;
CvSet* nv_heap;
CvRNG rng;
};
この構造体は,単独動作可能な決定木と決定木アンサンブルの両方を効率的に格納するために,主に内部的に利用されます.基本的に 3 種類の情報を含みます.
- 学習パラメータ.つまり CvDTreeParams のインスタンス.
- より効率的に最適な分岐を求めるために前処理された学習データ.決定木アンサンブルの場合,この前処理済みデータがすべての木で再利用されます.さらに,アンサンブルのすべての木で共有される学習データの特徴(変数の型,クラス数,クラスラベルの圧縮マップなど)もここに格納されます.
- バッファ.つまり 木ノード,分岐,木のその他の要素のためのメモリストレージ.
この構造体の利用の仕方には 2 通りあります.単純な場合(例えば,単独動作可能な木,または Random Trees や Boosting のような,すぐに使える「ブラックボックス」的な決定木アンサンブル)では,その構造体を気にしたり,あるいは知る必要さえなく,必要な統計モデルを単に作成し,それを学習して利用するだけです.この場合, CvDTreeTrainData は内部的に作成,利用されます.しかし,独自の木アルゴリズムを利用する場合,あるいは別の高度な事例においては,この構造体が明示的に作成・利用される場合もあります.その概要は以下のようになります:
- この構造体はデフォルトコンストラクタにより初期化された後 set_data を呼び出します(または,完全にパラメータを埋めるコンストラクタにより構成されます).パラメータ _shared は, true にセットされなければいけません.
- このデータを用いて,1つ以上の木が学習されます. CvDTree::train メソッドの特別な形式を参照してください.
- 最後に,この構造体を利用する木がすべて解放された後に,この構造体自身が解放されます.
CvDTree¶
- CvDTree¶
決定木
class CvDTree : public CvStatModel
{
public:
CvDTree();
virtual ~CvDTree();
virtual bool train( const CvMat* _train_data, int _tflag,
const CvMat* _responses, const CvMat* _var_idx=0,
const CvMat* _sample_idx=0, const CvMat* _var_type=0,
const CvMat* _missing_mask=0,
CvDTreeParams params=CvDTreeParams() );
virtual bool train( CvDTreeTrainData* _train_data,
const CvMat* _subsample_idx );
virtual CvDTreeNode* predict( const CvMat* _sample,
const CvMat* _missing_data_mask=0,
bool raw_mode=false ) const;
virtual const CvMat* get_var_importance();
virtual void clear();
virtual void read( CvFileStorage* fs, CvFileNode* node );
virtual void write( CvFileStorage* fs, const char* name );
// 決定木アンサンブルの木に対する,専用の read & write メソッド
virtual void read( CvFileStorage* fs, CvFileNode* node,
CvDTreeTrainData* data );
virtual void write( CvFileStorage* fs );
const CvDTreeNode* get_root() const;
int get_pruned_tree_idx() const;
CvDTreeTrainData* get_data();
protected:
virtual bool do_train( const CvMat* _subsample_idx );
virtual void try_split_node( CvDTreeNode* n );
virtual void split_node_data( CvDTreeNode* n );
virtual CvDTreeSplit* find_best_split( CvDTreeNode* n );
virtual CvDTreeSplit* find_split_ord_class( CvDTreeNode* n, int vi );
virtual CvDTreeSplit* find_split_cat_class( CvDTreeNode* n, int vi );
virtual CvDTreeSplit* find_split_ord_reg( CvDTreeNode* n, int vi );
virtual CvDTreeSplit* find_split_cat_reg( CvDTreeNode* n, int vi );
virtual CvDTreeSplit* find_surrogate_split_ord( CvDTreeNode* n, int vi );
virtual CvDTreeSplit* find_surrogate_split_cat( CvDTreeNode* n, int vi );
virtual double calc_node_dir( CvDTreeNode* node );
virtual void complete_node_dir( CvDTreeNode* node );
virtual void cluster_categories( const int* vectors, int vector_count,
int var_count, int* sums, int k, int* cluster_labels );
virtual void calc_node_value( CvDTreeNode* node );
virtual void prune_cv();
virtual double update_tree_rnc( int T, int fold );
virtual int cut_tree( int T, int fold, double min_alpha );
virtual void free_prune_data(bool cut_tree);
virtual void free_tree();
virtual void write_node( CvFileStorage* fs, CvDTreeNode* node );
virtual void write_split( CvFileStorage* fs, CvDTreeSplit* split );
virtual CvDTreeNode* read_node( CvFileStorage* fs,
CvFileNode* node,
CvDTreeNode* parent );
virtual CvDTreeSplit* read_split( CvFileStorage* fs, CvFileNode* node );
virtual void write_tree_nodes( CvFileStorage* fs );
virtual void read_tree_nodes( CvFileStorage* fs, CvFileNode* node );
CvDTreeNode* root;
int pruned_tree_idx;
CvMat* var_importance;
CvDTreeTrainData* data;
};
CvDTree::train¶
- bool CvDTree::train(const CvMat* _train_data, int _tflag, const CvMat* _responses, const CvMat* _var_idx=0, const CvMat* _sample_idx=0, const CvMat* _var_type=0, const CvMat* _missing_mask=0, CvDTreeParams params=CvDTreeParams())¶
- bool CvDTree::train(CvDTreeTrainData* _train_data, const CvMat* _subsample_idx)
決定木を学習します.
CvDTree には,2つの train メソッドが存在します.
第1形式のメソッドは,一般的な CvStatModel::train メソッドの形式に従います.これは,最も完全な形式です.ここでは,サンプルと変数の部分集合,データ欠損,入力変数と出力変数の型の任意の組み合わせなどに加えて,両方のデータレイアウト( _tflag=CV_ROW_SAMPLE と _tflag=CV_COL_SAMPLE )がサポートされます.最後のパラメータは,必要な学習パラメータをすべて含みます. CvDTreeParams の説明を参照してください.
第2形式のメソッドは,主に決定木アンサンブルを構築するために利用されます.これは,あらかじめ作成された CvDTreeTrainData インスタンスと,オプションとして学習データの部分集合を引数にとります. _subsample_idx は, _sample_idx の値を指すインデックスとみなされ, CvDTreeTrainData コンストラクタに渡されます.例えば _sample_idx=[1, 5, 7, 100] の場合, _subsample_idx=[0,3] は,元の学習データ集合内のサンプル [1, 100] が利用されることを意味します.
CvDTree::predict¶
- CvDTreeNode* CvDTree::predict(const CvMat* _sample, const CvMat* _missing_data_mask=0, bool raw_mode=false) const¶
入力ベクトルに対する,決定木の葉ノードを返します.
このメソッドは,特徴ベクトルと,オプションとしてデータ欠損マスクを引数に取り,決定木を辿って到達した葉ノードを出力として返します.予測結果,つまりクラスラベル,または推定された関数の値が, CvDTreeNode 構造体の value フィールド値として(例えば, dtree->predict(sample,mask)->value というように)読み出せるようになります.
最後のパラメータは通常,通常の入力を意味する
false
にセットされます.これが
true
の場合,このメソッドは,離散入力変数のすべての値が,予め
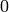 から
から
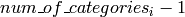 の範囲に正規化されている,と仮定します(決定木は,このような正規化された表現を内部的に利用しています).この場合,決定木アンサンブルの予測がより高速になります.また,入力が連続変数の場合,このフラグは利用されません.
の範囲に正規化されている,と仮定します(決定木は,このような正規化された表現を内部的に利用しています).この場合,決定木アンサンブルの予測がより高速になります.また,入力が連続変数の場合,このフラグは利用されません.
例:キノコを分類する決定木. 決定木をどのように構築し,利用するかを実演してくれる mushroom.cpp サンプルを参照してください.
ヘルプとフィードバック
お探しの情報が見つかりませんか?- チーシート(PDF) を読んでみてください.
- ユーザグループ/メーリングリスト (英語)で質問してみてください.
- このドキュメントに誤りなどを見つけたらbug report まで報告してください.また,日本語訳に関する誤りや指摘はOpenCV.jpまでお願いします.
