ランダムツリー¶
ランダムツリーは Leo Breiman と Adele Cutler によって発表されました: http://www.stat.berkeley.edu/users/breiman/RandomForests/ . このアルゴリズムは,分類問題と回帰問題の両方を扱うことができます.ランダムツリーは,木構造を持つ予測器のコレクション(アンサンブル)であり,このセクションではこれを forest (この用語も L. Breiman によるものです)と呼びます.この分類器は,次のように動作します:ランダムツリー分類器は,入力特徴ベクトルを取り込んで forest 内のすべての木によりその分類を行い,その多くの「投票」を得たクラスラベルを出力します.また回帰の場合,分類器の応答は,forest 内のすべての木の応答の平均値となります.
すべての木は同じパラメータで学習されますが,そこで用いられるデータ集合はブートストラップ法によって元の学習データから生成され,それぞれの木で異なります:各学習データとして,同じ個数のベクトルを元の集合(
=N
)からランダムに選択します.ここで,ベクトルは置き換えにより選択されます.つまり,複数回使用されるベクトルもあれば,まったく使用されないベクトルもあります.学習される木のそれぞれのノードにおける最適な分岐を求めるために,すべての変数ではなく,ここからランダムに選択された部分集合が利用されます.それぞれのノードで新しい部分集合が生成されますが,そのサイズはすべてのノードおよびすべての木について固定値です.これは学習パラメータであり,デフォルトでは
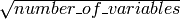 となります.また,構成された木の枝刈りは行われません.
となります.また,構成された木の枝刈りは行われません.
ランダムツリーでは,交差検証法やブートストラップ法のように精度を推定する必要はなく,学習誤差を推定するための個別のテスト集合も不要です.この誤差は,学習時に内部で推定されます.現在の木に対する学習データ集合が,データの置き換えを伴うサンプリングで抽出される際に,いくつかのベクトルが除外されます(いわゆる oob (out-of-bag) データ です).oobデータのサイズは,約 N/3 です.そして分類誤差は,この oobデータを用いて次のように推定されます:
- i番目の木を利用して,oobデータの各ベクトル(これは相対的にi番目の木に対してもoobデータになります)に対する予測を行います.
- すべての木の学習が終了した後で,oobデータであった各ベクトルに対する「勝者」クラス(つまり,oobデータを入力ベクトルとした場合に,最も多くの投票を得たクラス)を求めます.そして,それを真値と比較します.
- そして,oobベクトルを誤分類した数と元データの全ベクトル数との比率として,分類誤差を計算します.また,回帰の場合のoob誤差は,oobベクトルに対する予測の和をベクトルの総数で割った値と真値との平方誤差として計算されます.
参考文献:
Machine Learning, Wald I, July 2002.
Looking Inside the Black Box, Wald II, July 2002.
Software for the Masses, Wald III, July 2002.
And other articles from the web site http://www.stat.berkeley.edu/users/breiman/RandomForests/cc_home.htm .
CvRTParams¶
- CvRTParams¶
ランダムツリーの学習パラメータ.
struct CvRTParams : public CvDTreeParams
{
bool calc_var_importance;
int nactive_vars;
CvTermCriteria term_crit;
CvRTParams() : CvDTreeParams( 5, 10, 0, false, 10, 0, false, false, 0 ),
calc_var_importance(false), nactive_vars(0)
{
term_crit = cvTermCriteria( CV_TERMCRIT_ITER+CV_TERMCRIT_EPS, 50, 0.1 );
}
CvRTParams( int _max_depth, int _min_sample_count,
float _regression_accuracy, bool _use_surrogates,
int _max_categories, const float* _priors,
bool _calc_var_importance,
int _nactive_vars, int max_tree_count,
float forest_accuracy, int termcrit_type );
};
forest の学習パラメータは,単一の木の学習パラメータの上位集合となります.しかし,ランダムツリーは,決定木の機能や特徴をすべて必要とするわけではありません.その最たる違いとして,木は枝刈りされません.よって,交差検証法のパラメータも利用されません.
CvRTrees¶
- CvRTrees¶
ランダムツリー.
class CvRTrees : public CvStatModel
{
public:
CvRTrees();
virtual ~CvRTrees();
virtual bool train( const CvMat* _train_data, int _tflag,
const CvMat* _responses, const CvMat* _var_idx=0,
const CvMat* _sample_idx=0, const CvMat* _var_type=0,
const CvMat* _missing_mask=0,
CvRTParams params=CvRTParams() );
virtual float predict( const CvMat* sample, const CvMat* missing = 0 )
const;
virtual void clear();
virtual const CvMat* get_var_importance();
virtual float get_proximity( const CvMat* sample_1, const CvMat* sample_2 )
const;
virtual void read( CvFileStorage* fs, CvFileNode* node );
virtual void write( CvFileStorage* fs, const char* name );
CvMat* get_active_var_mask();
CvRNG* get_rng();
int get_tree_count() const;
CvForestTree* get_tree(int i) const;
protected:
bool grow_forest( const CvTermCriteria term_crit );
// forest を構成する木の配列
CvForestTree** trees;
CvDTreeTrainData* data;
int ntrees;
int nclasses;
...
};
cv::CvRTrees::train¶
- bool CvRTrees::train(const CvMat* train_data, int tflag, const CvMat* responses, const CvMat* comp_idx=0, const CvMat* sample_idx=0, const CvMat* var_type=0, const CvMat* missing_mask=0, CvRTParams params=CvRTParams())¶
- ランダムツリーモデルの学習を行います.
CvRTrees::train メソッドは, CvDTree::train () の最初の形式ととてもよく似ており,一般的な CvStatModel::train 形式に従います.このアルゴリズムの学習パラメータの指定はすべて, CvRTParams のインスタンスという形で渡されます.また,学習誤差( oob-誤差 )の推定値は,プロテクトメンバ obb_error に格納されます.
cv::CvRTrees::predict¶
この予測メソッドの引数は, CvDTree::predict のものと同じですが,戻り値の型は異なります.このメソッドは,forest のすべての木の累積結果(多数が指示するクラス,または回帰関数の推定値の平均)を返します.
cv::CvRTrees::get_var_importance¶
このメソッドは, :ref:`CvRTParams`::calc_var_importance がセットされている場合に学習時に計算される,変数の重要度のベクトルを返します.この学習フラグがセットされていない場合, NULL ポインタが返されます.また,決定木とは異なり,学習後にいつでも変数の重要度を計算することができます.
cv::CvRTrees::get_proximity¶
- float CvRTrees::get_proximity(const CvMat* sample_1, const CvMat* sample_2) const¶
- 2つの学習サンプルの近接度を取得します.
このメソッドは,任意の2つのサンプルの近接度(2つのサンプルが同じ葉ノードに到達するような木の個数の,アンサンブルの木の総数に対する割合)を返します.
例:ランダムツリー分類器を用いた,毒キノコの判定
#include <float.h>
#include <stdio.h>
#include <ctype.h>
#include "ml.h"
int main( void )
{
CvStatModel* cls = NULL;
CvFileStorage* storage = cvOpenFileStorage( "Mushroom.xml",
NULL,CV_STORAGE_READ );
CvMat* data = (CvMat*)cvReadByName(storage, NULL, "sample", 0 );
CvMat train_data, test_data;
CvMat response;
CvMat* missed = NULL;
CvMat* comp_idx = NULL;
CvMat* sample_idx = NULL;
CvMat* type_mask = NULL;
int resp_col = 0;
int i,j;
CvRTreesParams params;
CvTreeClassifierTrainParams cart_params;
const int ntrain_samples = 1000;
const int ntest_samples = 1000;
const int nvars = 23;
if(data == NULL || data->cols != nvars)
{
puts("Error in source data");
return -1;
}
cvGetSubRect( data, &train_data, cvRect(0, 0, nvars, ntrain_samples) );
cvGetSubRect( data, &test_data, cvRect(0, ntrain_samples, nvars,
ntrain_samples + ntest_samples) );
resp_col = 0;
cvGetCol( &train_data, &response, resp_col);
/* 行列 missed の作成 */
missed = cvCreateMat(train_data.rows, train_data.cols, CV_8UC1);
for( i = 0; i < train_data.rows; i++ )
for( j = 0; j < train_data.cols; j++ )
CV_MAT_ELEM(*missed,uchar,i,j)
= (uchar)(CV_MAT_ELEM(train_data,float,i,j) < 0);
/* comp_idx ベクトルの作成 */
comp_idx = cvCreateMat(1, train_data.cols-1, CV_32SC1);
for( i = 0; i < train_data.cols; i++ )
{
if(i<resp_col)CV_MAT_ELEM(*comp_idx,int,0,i) = i;
if(i>resp_col)CV_MAT_ELEM(*comp_idx,int,0,i-1) = i;
}
/* sample_idx ベクトルの作成 */
sample_idx = cvCreateMat(1, train_data.rows, CV_32SC1);
for( j = i = 0; i < train_data.rows; i++ )
{
if(CV_MAT_ELEM(response,float,i,0) < 0) continue;
CV_MAT_ELEM(*sample_idx,int,0,j) = i;
j++;
}
sample_idx->cols = j;
/* マスクの作成 */
type_mask = cvCreateMat(1, train_data.cols+1, CV_8UC1);
cvSet( type_mask, cvRealScalar(CV_VAR_CATEGORICAL), 0);
// 学習パラメータの初期化
cvSetDefaultParamTreeClassifier((CvStatModelParams*)&cart_params);
cart_params.wrong_feature_as_unknown = 1;
params.tree_params = &cart_params;
params.term_crit.max_iter = 50;
params.term_crit.epsilon = 0.1;
params.term_crit.type = CV_TERMCRIT_ITER|CV_TERMCRIT_EPS;
puts("Random forest results");
cls = cvCreateRTreesClassifier( &train_data,
CV_ROW_SAMPLE,
&response,
(CvStatModelParams*)&
params,
comp_idx,
sample_idx,
type_mask,
missed );
if( cls )
{
CvMat sample = cvMat( 1, nvars, CV_32FC1, test_data.data.fl );
CvMat test_resp;
int wrong = 0, total = 0;
cvGetCol( &test_data, &test_resp, resp_col);
for( i = 0; i < ntest_samples; i++, sample.data.fl += nvars )
{
if( CV_MAT_ELEM(test_resp,float,i,0) >= 0 )
{
float resp = cls->predict( cls, &sample, NULL );
wrong += (fabs(resp-response.data.fl[i]) > 1e-3 ) ? 1 : 0;
total++;
}
}
printf( "Test set error =
}
else
puts("Error forest creation");
cvReleaseMat(&missed);
cvReleaseMat(&sample_idx);
cvReleaseMat(&comp_idx);
cvReleaseMat(&type_mask);
cvReleaseMat(&data);
cvReleaseStatModel(&cls);
cvReleaseFileStorage(&storage);
return 0;
}
ヘルプとフィードバック
お探しの情報が見つかりませんか?- FAQ (英語)を試してください.
- ユーザグループ/メーリングリスト (英語)で質問してみてください.
- このドキュメントに誤りなどを見つけたらbug report まで報告してください.また,日本語訳に関する誤りや指摘はopencv.jpまでお願い します.
